Pythonの高速化について
この記事では,Pythonの実行高速化手法について書いていきます.
あくまでも個人の意見であり,どの程度正しいかわからないので適宜調べてください.
今回は主に
- プロファイリングによる高速化(cProfile)
- NumPyにおける高速化
- コンパイルによる高速化(Cython, Numba)
について書いていこうと思います.
個人的な意見ですが,優先度は
プロファイリング >> (使ってるならNumPy) >> コンパイル
だと思ってます.
遅いなと思ったらとりあえずプロファイリングしましょう.
プロファイリングによる高速化(cProfile)
書いてある通り,cProfileモジュールを使ってプロファイリングをします.
プロファイリングというのは,ソースコードを実行したときにどの関数が何回実行されているか,実行ごとにどのくらいの時間がかかっているかなどの情報を得ることで,これをすることで時間がかかっている処理を特定することができます.
前に書いた記事でも名前だけ出している方法です.
toki0177.hatenablog.com
パレートの法則の例としてよく言われる「プログラムの処理にかかる時間の80%はコード全体の20%の部分が占める。」というものがあります.
これはつまりコード全体の20%分について実行時間を減らすことができれば大幅な高速化が見込めるということです.
それではやっていきましょう!
プロファイリングを行うコードは以下のようになっています.
import cProfile import pstats pr = cProfile.Profile() pr.enable() # ここにプロファイリングしたい処理を書く pr.disable() stats = pstats.Stats(pr) stats.sort_stats('cumtime') stats.print_stats() pr.dump_stats('profile.stats')
ここでの処理の流れは
- Profileクラスのインスタンスを作成
- enableでプロファイリングを開始
- disableでプロファイリングを終了
- cumtime(総実行時間)で並び替え
- プロファイリング結果の出力
- ファイルに書き出し
のようになっています.
enableとdisableの間に記述した処理について実行時間などの計測を行います.
プロファイル結果において注目すべきは総実行時間や呼び出し回数,呼び出しごとの実行時間などいろいろなものがありますが,その時々で適切な部分を見ましょう.
これを実行すると,以下のような出力がされると思います.
Ordered by: cumulative time
ncalls tottime percall cumtime percall filename:lineno(function)
130/1 0.004 0.000 22.159 22.159 {built-in method builtins.exec}
1 0.000 0.000 22.159 22.159 hoge.py:1(<module>)
1 0.001 0.001 21.937 21.937 hoge.py:12(main)
1 0.017 0.017 9.810 9.810 hoge.py:154(hoge1)
11 4.256 0.387 9.397 0.854 hoge.py:175(hoge2)
65667 1.524 0.000 4.314 0.000 hoge.py:149(hoge3)
131334 0.092 0.000 2.066 0.000 {method 'sum' of 'numpy.ndarray' objects}
161078 2.059 0.000 2.059 0.000 {method 'reduce' of 'numpy.ufunc' objects}これはある程度実行時間を短縮した後のものなのでここから変更するべき部分はあまりないかもしれませんが,実行時の様子を調べることができます.
今回は実行時間について調べましたが,その他にも
- print_callers() : 呼び出した関数の情報
- print_callees() : 呼び出された関数の情報
なども調べることができます.
このプロファイリングの結果を見て,実行が多すぎる部分を調べたり,1回ごとの実行時間が長い処理を調べてそこの実行時間を短縮すればいいということになります.
次に,プロファイリングの結果で「NumPyを使ってる部分の処理に時間がかかっている!」というときの対処について考えていきます.
NumPyにおける高速化
ここではNumPyにおける高速化の例を示していきます.
最も肝心なのはfor文をできる限り使わないことです.
たぶんどのページ見ても同じようなことを言われているとは思いますが,Pythonのfor文は遅いです.
NumPyはfor文を用いなくてもほとんどの処理ができると思っているので,できる限りfor文を使わない表記にしましょう.
例えば下のような関数を書いたとします.
適当に作りましたが2つの画像の各画素のユークリッド距離の総和を求める関数です.
def calc_distance(img1, img2): tmp = cv2.absdiff(img1, img2).astype(np.uint64) ret = 0 for i in range(len(tmp)): for j in range(len(tmp[i])): ret += np.sqrt(tmp[i,j,0]**2+tmp[i,j,1]**2+tmp[i,j,2]**2) return ret
このコードをtimeitで計測した結果は以下の通りです.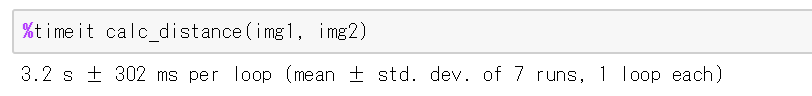
次に,for文を無くして高速化したコードは以下の通りです.
def calc_distance(img1, img2): tmp = cv2.absdiff(img1, img2).astype(np.uint64) return np.sqrt((tmp ** 2).sum(2)).sum()
行っている処理の内容はほぼ同じですが,timeitで計測した結果は以下の通りです.
このようにNumPyではfor文を使わなくても処理ができることが多いので,できる限りfor文を使わないようにしましょう.
コンパイルによる高速化(Cython, Numba)
これについてはあまり詳しくは書きません.
調べるとたくさん出てくるので調べましょう.
これで高速化できる場合とあまり効果がない場合がある気がします.
簡単に言うと,Pythonは1行ずつ翻訳して実行していく(たぶん)のですが,コンパイルして実行するC言語などと比較すると低速です.
そこで,コンパイルをして実行を高速化しようとするという考えで作られたのがCythonとNumbaです(たぶん).
Cythonは実行前にコンパイルし,Numbaは実行時にコンパイルみたいな感じだと思います.
Pythonの実行が遅い理由の一つとして言われるのが型の宣言をしないので参照のときに型を調べる必要がありますが,コンパイルによる高速化以外にも明示的な型宣言をすることで高速化をすることもできます.
まとめ
とりあえずプロファイリングをしましょう.
今回言いたかったのはだいたいそれです.